Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Jornal Português de Gastrenterologia
Print version ISSN 0872-8178
J Port Gastrenterol. vol.18 no.5 Lisboa Sept. 2011
Genotipagem do Helicobacter pylori no Carcinoma Gástrico e Gastrite Crónica
Maria Reis Silva1,2, Carla Oliveira2, Paulo Rodrigues-Santos2,3, Lina Carvalho1
1Instituto de Anatomia Patológica, Faculdade de Medicina da Universidade de Coimbra, 2Escola Superior de Tecnologia da Saúde de Coimbra, 3Instituto de Imunologia, Faculdade de Medicina da Universidade de Coimbra
RESUMO
A infecção gástrica provocada pelo Helicobacter pylori (Hp) é a principal causa da gastrite crónica do tipo B e condição pré neoplásica para o desenvolvimento de carcinoma gástrico e linfoma MALT. A patogenicidade provocada por diferentes estirpes do Hp está associada à função de alguns genes desta bactéria e respectivos oligonucleotídeos, como o cagA, vacA e iceA. Pretendeu-se pesquisar a presença destes genes em produtos de biopsia de carcinoma gástrico e gastrite crónica. O DNA bacteriano foi extraído de 39 amostras fixadas em formol e incluídas em parafina, das quais 26 correspondiam a carcinoma gástrico e 13 a gastrite crónica, por 2 técnicas distintas: secções das biopsias sobre lâminas e colhidas para tubo com xilol. Os genes cagA, vacA e iceA foram depois pesquisados através de nested-PCR. Do estudo de associações entre os genótipos das estripes de H. pylori em CG e GC encontrou-se diferenças estatisticamente significativas para o alelo ice A1 (p < 0,05; OR = 4,25). A maioria dos GC com cagA+ estão associadas ao vacA s2/m2 (46%,12/26). A combinação s2/m2/cagA/iceA1 ocorreu predominantemente em 35% das CG comparado com 30% dos GC, (P > 0.05; OR = 1,19). Para as combinações s2/m2/cagA/iceA2 e s1/m2/cagA/ice2 obteve-se um valor de 12% para CG e 30% para GC, (P > 0,05; OR = 0,29). Estes conhecimentos são importantes para continuidade de outros estudos que pretendam determinar as características do genótipo de H. pylori nas lesões pré-neoplásicas do estômago.
Palavras-chave:Helicobacter pylori; carcinoma gástrico; gastrite crónica; cagA; vacA; iceA; nested PCR.
Genotypes of Helicobacter pylori in Gastric Cancer and Chronic Gastritis
ABSTRACT
Helicobacter pylori (Hp) infection is the major cause of chronic active gastritis type B and a pre-neoplasic condition to the development of gastric carcinoma and MALT lymphoma. The pathogenicity induced by different Hp strains are associated to the functions of some Hp genes and respective oligonucleotides, such as cagA, vacA and iceA. This study was designed to assemble these genes in gastric carcinoma and chronic gastritis biopsies. The Hp DNA was extracted from 39 formalin fixed paraffin embedded gastric samples, 26 from gastric carcinoma and 13 from chronic gastritis through 2 different methods: sections on glass slides and isolated sections cut to eppendorf tubes. The cagA, vacA and iceA genes were then detected by nested-PCR. The study of associations between the genotypes of the strains of H. pylori in GC and CG was found statistically significant differences for allele ice A1 (p < 0.05, OR = 4.25). Most of the GC with cagA + vacA are associated with s2/m2 (46%, 12/26). The combination s2/m2/cagA/iceA1 predominantly occurred in 35% of CG compared with 30% of GC (P > 0.05, OR = 1.19). For combinations s2/m2/cagA/iceA2 and s1/m2/cagA/ice2 obtained a value of 12% and 30% for GC and GC (P> 0.05, OR = 0.29). This knowledge is important for continuity of further studies aiming to define the characteristics of H. pylori genotype in pre-neoplasic lesions of the stomach.
Keywords:Helicobacter pylori; gastric carcinoma; chronic gastritis; cagA; vacA; iceA; nested-PCR.
INTRODUÇÃO
O Helicobacter pylori (Hp) é um bacilo Gram negativo e microaerofílico, da família Enterobacteriaceae, capaz de colonizar a superfície da mucosa gástrica e persistir durante décadas no estômago, na ausência de tratamento farmacológico apropriado1-4. Esta bactéria é considerada o principal agente etiológico da gastrite crónica (GC) do tipo B e também da úlcera duodenal (UD), do carcinoma gástrico (CG) e do linfoma de MALT (LM)3,4. O mecanismo de colonização caracteriza-se pela penetração da bactéria nas criptas do epitélio superficial do estômago, seguindo-se a adaptação ao meio e posterior aderência2,3. A sua principal característica é a sobrevivência num meio ácido e tem a capacidade de alterar o pH do estômago através da produção da enzima urease1,5,6 induzindo hipocloridria progressiva e atrofia gástrica5.
A infecção causada pelo Hp tem sido considerada uma das mais frequentes e principais patologias bacterianas crónicas nos seres humanos, dado que estudos recentes demonstraram que cerca de 50% da população mundial apresenta colonização gástrica3,7. Contudo, verifica-se que a maioria das pessoas são assintomáticas, encontrando-se apenas uma pequena percentagem (15 a 20) de indivíduos que apresentam sintomas8-10, havendo uma maior prevalência da infecção nos países em vias de desenvolvimento, onde os hábitos de higiene e de alimentação são deficientes; os factores ambientais e sócio-económicos são determinantes para o seu aparecimento e posterior manutenção11,12.
Muitos estudos foram desenvolvidos para esclarecer a razão pela qual apenas uma minoria da população com esta infecção desenvolve o CG e quais os genes do Hp que estão implicados11,12. A patogenicidade de Hp está associada à composição do genoma da bactéria e este pode ser correlacionado com a severidade das lesões gástricas induzidas8,13,14. Neste contexto, tem vindo a ser estudada a importância da associação de alguns genes de Hp, tais como o cagA, o vacA e o iceA, com as lesões gástricas e a sua manutenção ou evolução11,14,15.
O gene cagA foi o primeiro a ser identificado11,14,16 e codifica a proteína CagA – cytotoxin associated gene A. Este gene pertence a uma região do genoma bacteriano com 40 Kb contendo 31 genes, denominada por ilhéu de patogenicidade (PAI) cag16,17. A presença desta região caracteriza estirpes de Hp que apresentam um maior grau de virulência – Tipo I, enquanto que a sua ausência está associada a um grupo de estirpes com menor grau de virulência – tipo II18,19. É importante referir que a proteína CagA é produzida por 60% das estirpes de Hp, sendo responsável por um aumento da resposta inflamatória gástrica, pois promove a libertação de citocinas pró-inflamatórias, tais como, IL-820. A proteína é libertada no citoplasma da célula epitelial gástrica, sendo posteriormente fosforilada nos resíduos tirosina-cinase que induz a secreção da IL-8, um potente quimiotático e factor activador dos neutrófilos. A proteína CagA também é capaz de induzir a transcrição de vários proto-oncogenes, tais como, c-fos ou c-jun através, por exemplo, da cascata das MAP cinases5. Sendo assim, os eventos anteriormente descritos são normalmente observados quando se desenvolveram lesões pré-neoplásicas que estão relacionadas com a presença do gene cag A5,17.
O gene vacA codifica a proteína VacA, vacuolating cytotoxin, que promove a formação de vacúolos nas células epiteliais gástricas, assim como o aumento da permeabilidade celular e apoptose epitelial, conduzindo à ulceração e, consequentemente, ao desenvolvimento de lesões pré-neoplásicas e do CG7. A expressão deste gene é determinada por variações na sequência de nucleótidos, nomeadamente nas regiões de sinal – alelos s1 e s2 – e média – alelos m1 e m2. A produção das regiões do gene vacA está relacionada com a combinação das mesmas, sendo que: a combinação s1/m1 associa-se a estirpes muito virulentas e a combinação s1/m2 associa-se a estirpes com virulência moderada; a combinação s2/m2 verifica-se em estirpes pouco virulentas21.
Por último, o gene IceA codifica a proteína IceA – induced by contact with epithelium também produzida após o contacto da bactéria com o epitélio gástrico24. O gene iceA tem duas variantes alélicas: o iceA1 e o iceA222,23. Ainda é desconhecida a função exacta dos produtos das mesmas e sabe-se que a variante iceA1 está associada a um aumento da concentração de IL-8, no desenvolvimento da úlcera duodenal, e apresenta homologia com a endonuclease de restrição NlaIII da Neisseria lactamica. Por outro lado, a variante iceA2 está associada à severidade da inflamação em determinados locais geográficos. Deste modo, o gene iceA é um importante marcador de elevada patogenicidade do Hp, estando associado ao desenvolvimento de condições gástricas pré-neoplásicas mais severas5,23,24.
Assim, de acordo com os conhecimentos actuais, novas terapias podem surgir e prevenir, ou mesmo impedir, o desenvolvimento de neoplasias gástricas malignas provocadas por esta bactéria25,26.
Neste estudo, foi explorada a nested PCR como técnica de eleição para identificação dos genes cagA, vacA, iceA e determinar diferentes genótipos na GC e no CG. A utilização da nested PCR fornece a possibilidade de trabalhar com biopsias gástricas previamente fixadas em formol e incluídas em parafina, excluindo a necessidade do estudo de estirpes do Hp a partir de culturas microbiológica. Pretendeu-se assim, identificar e estabelecer uma relação entre os diferentes genótipos de Hp com o Carcinoma Gástrico e com a Gastrite Crónica.
MATERIAL E MÉTODOS
Material
Foram estudadas biopsias gástricas fixadas em formol e incluídas em parafina, do arquivo do Instituto de Anatomia Patológica da Faculdade de Medicina da Universidade de Coimbra de 2005. A cada amostra correspondia um relatório histológico, tendo sido o Hp previamente identificado pela técnica de Giemsa modificado, orientando a sua inclusão no estudo.
Histopatologia
A população estudada compreendeu 19 indivíduos do género masculino e 20 indivíduos do género feminino sendo que 30 pacientes estavam diagnosticados com CG (idade média, 67,2 ± 12,7 anos), dos quais 9 tinham carcinoma gástrico do tipo difuso, 15 carcinomas gástricos do tipo intestinal e 2 carcinomas gástricos do tipo indeterminado, de acordo com a classificação de Laurén. Nos restantes 13 pacientes foi diagnosticada GC (idade média, 48,7 ± 15,9anos) pela classificação de Sidney.
Secções de 4 μ m foram obtidos e corados com Hematoxilina - Eosina, Alcian blue - ácido periódico de Schiff (pH 2,5) e Giemsa. Dois patologistas (LC e MRS) reviram as biopsias iniciais e final de todos os casos.
MÉTODOS
Métodos
Extracção de DNA e PCR
Foram utilizados 2 métodos distintos para extracção de DNA a partir das biopsias gástricas incluídas em parafina.
De acordo com o protocolo de extracção do QIAamp DNA Mini Kit da QIAGEN, procedeu-se ao corte de 5 a 8 secções com cerca de 10 micra de espessura de cada bloco de parafina, tendo sido posteriormente adicionado xilol. Após centrifugação, as secções foram lavadas com etanol a 96-100%, seguindo-se a lise da parede bacteriana e purificação do DNA obtido.
Por outro lado, de acordo com o protocolo de extracção de DNA de tecido sobre lâmina, procedeu-se ao corte de 4 secções de cada amostra, com 8 - 10µm de espessura cada, colocando-as nas respectivas lâminas deixadas na estufa a 37ºC de um dia para o outro. Cada lâmina foi desparafinizada com xilol, seguindo-se várias passagens com álcool a 100%, 90% e 70%. Após microdissecção manual do tecido contido na lâmina, incubou-se o material a 55ºC, com a seguinte mistura: 50mM Tris HCl, 1mM EDTA, 1% Tween 20 e 3mg/ml Proteinase K (Roche; Basel, Suíça). Colocou-se os tubos a 100ºC durante 10 minutos, de modo a inactivar a proteinase K, introduzindo-os logo de seguida em gelo durante 10 minutos.
Para pesquisa dos genes cagA, vacA e iceA foram seleccionados primers idênticos às sequências nucleotídicas usadas por Koehler et al., publicados no Genbank database do NCBI e criados por MWG-Biotech (Ebersberg, Alemanha).
A partir de cada set de primers foi realizada uma PCR nested, excepto na pesquisa das regiões s1 e s2 do gene vacA em que foi realizado uma semi-nested PCR, obtendo-se fragmentos de 102 pb e 301 pb.
A primeira mistura I para a PCR consistiu na adição da solução de trabalho, (50mM MgCl2, 10x tampão NH4, 10mM dNTPs, 99% glicerol, 10 mg/ml vermelho de cresol e água destilada), DNA (100-200 mg/µl), primers (após optimização 3,3mM do primer directo a 6,7mM do primer reverso) e 1 Unidade de Taq DNA polimerase (Roche, Basel, Suíça). Programa num termociclador (Biometra, UNO II) pré-incubação a 95ºC durante 5 minutos, seguindo-se 41 ciclos de desnaturação a 95ºC/1 minuto, 58ºC/1 minuto e 72ºC/1 minuto, finalizando com uma última extensão a 72ºC, durante 5 minutos. Após esta primeira amplificação, procedeu-se a uma segunda PCR na qual se utilizou 1µl do DNA amplificado com primers para o PCR nested, repetindo os passos do protocolo anteriormente descrito. O perfil electroforético de cada amostra foi observado num gel TAE de agarose a 2,5%.
ANÁLISE ESTATÍSTICA
A relação dos diferentes genótipos das várias estirpes do Hp identificados, com a severidade da infecção, na GC e com o GC, foi analisada através do teste do Qui-Quadrado (χ2) e do cálculo do Odds Ratio (OR) com intervalo de confiança de 95% (95% IC).
RESULTADOS
Electroforese
Após a electroforese, foram observados fragmentos de vários tamanhos, nomeadamente produtos de 120 pb para vacA s1, 150 pb para vacA s2, 301 pb para vacA m1, 102 pb para vacA m2, 181 pb para cagA, 218 pb para iceA1 e 247 pb para iceA2 (Fig. 1).
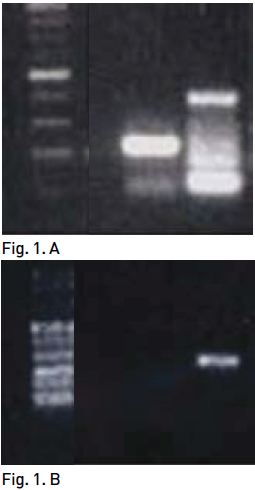
Fig. 1.Observação electroforética de alguns genes/regiões do H. pylori. (A) Presença da região vacA m2 (102 pb) e do gene cagA (181 pb). (B) Presença da região vacA s1 (150 pb) e da variante iceA1 (218 pb).
Distribuição dos genes cagA, vacA, iceA e respectivos alelos no CG e na GC
De acordo com os resultados obtidos nas 26 amostras diagnosticadas como CG e nas 13 amostras diagnosticadas como GC, foi possível realizar uma análise estatística através dos testes do Qui-Quadrado (χ2) e do Odds Ratio (OR) (Quadro 1).
Quadro 1. Distribuição dos genes cagA, vacA, iceA e seus respectivos alelos pelas biopsias de CG e GC e análise estatística dos resultados obtidos
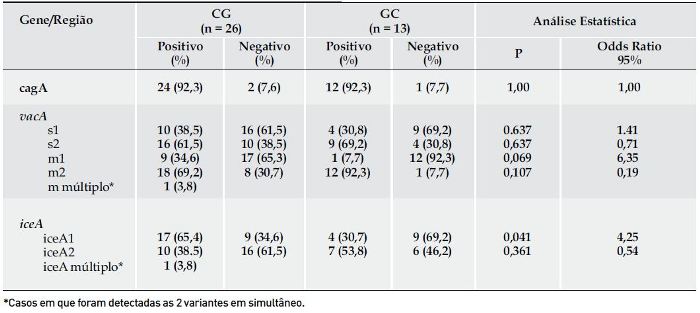
O gene cagA foi encontrado em 92,3% dos pacientes com GC e CG.
A região vacA s1 foi detectada em 38,5% dos CG e em 30,8% das GC (P > 0.05; OR = 1,41), ou seja, o risco de se encontrar este alelo no CG foi maior do que na GC. Em contraste, a região vacA s2 foi detectada em 61,5% dos CG e em 69,2% das GC, (P > 0.05; OR = 0,71). A região vacA m1 foi detectada em 34,6% dos CG e em 7,7% das GC (P >0,05; OR=6,35). Por outro lado, a região vacA m2 foi encontrada em 69,2% dos CG e em 92,3% das GC, (P > 0.05; OR = 0,19).
A região iceA1 foi mais prevalente do que a região iceA2, sendo que iceA1 foi detectada em 65,4% dos CG e em 30,7% das GC, apresentando valores estatisticamente significativos com um P = 0,04 e um OR = 4,25, isto é, o risco de se encontrar esta variante no CG foi maior do que na GC. Já a região iceA2 foi detectada em 38,5% dos CG e em 58,8% das GC, (P > 0,05; OR = 0,54).
Foi encontrado um caso de CG em que foram detectadas ambas as regiões médias da região vacA 3,8%. O mesmo aconteceu noutro caso de CG em que foram detectadas ambas as variantes do gene iceA 3,8%.
Tendo em conta os valores obtidos no cálculo do OR verificou-se que, vacA s2, vacA m2 e iceA1 foram predominantes no CG, enquanto que vacA s2, vacA m2 e iceA2 foram predominantes na GC. O gene cagA prevaleceu em ambas as patologias.
Distribuição dos genótipos mais frequentes do H. pylori detectados no CG e na GC
Foram estudados os genótipos mais frequentes do H. pylori, considerados importantes factores de risco para o desenvolvimento de CG e GC (Fig. 2). Observou-se que: a combinação s2/m2/cagA/iceA1 ocorreu predominantemente em 35% das CG comparado com 30% dos GC, (P > 0,05; OR = 1,19). Para as combinações s2/m2/cagA/iceA2 e s1/m2/cagA/ice2 obteve-se um valor de 12% para CG e 30% para GC, (P0,05; OR = 0,29). Na segunda, obteve-se um valor de 8% para CG e 23% para GC, (P > 0,05; OR = 0,19). Em contraste, a combinação s1/m2/cagA/iceA1 ocorreu predominantemente em 23% dos CG comparado com um valor de 0% para GC (P > 0,05). A mesma situação foi observada para as combinações s1/m2/cagA/iceA1,2 e s1/m1/cagA/iceA2. A primeira, na presença das 2 regiões de iceA, apresentou um valor de 3,8% para CG e 0% para GC. A segunda apresentou um valor de 12% para CG e 0% para GC.
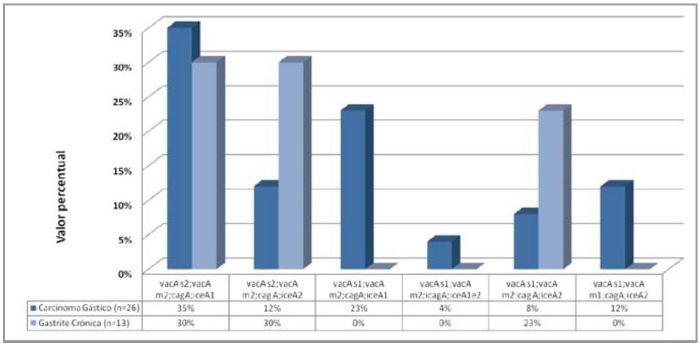
Fig. 2. Distribuição dos genótipos mais frequentemente encontrados em estirpes de H. pylori, após o estudo de biopsias de tecido com CG e GC.
A associação mais frequente no CG incluindo os três genótipos foi o cagA+/vacA s2/m2, ice A1 (35%), e para a GC cagA+/vacA s2/m2, ice A1 (30%) e cagA+/vaca s2/m2, ice A2 (30%).
Relação entre os vários tipos de CG e as diferentes combinações formadas pelas regiões de sinal e média do gene vacA e pelas variantes do gene iceA
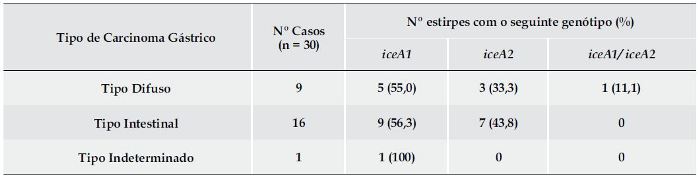
De acordo com o relatório histológico, os casos de CG foram distribuídos pelos três tipos histológicos da classificação de Laurén: intestinal, difuso e indeterminado (Quadro 2).
Quadro 2. Relação entre os vários tipos de CG e as diversas combinações das regiões de sinal e média do gene vacA.
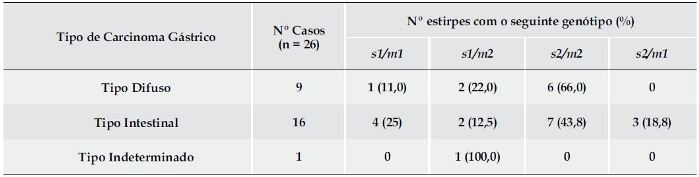
No que diz respeito às combinações entre as regiões de sinal e média do gene vaca, o genótipo s2/m2 ocorreu frequentemente no CG do tipo difuso (66%), seguindo-se o genótipo s1/m2 (22%) e o genótipo s1/m1 (11%), que foi o menos prevalente. O mesmo se verificou no CG do tipo intestinal onde a combinação s2/m2 foi a mais frequente (43,8%), seguindo-se as combinações s1/m1 com 25%, s1/m2 (12,5%) e s2/m1 (18,8%). No CG do tipo indeterminado foi detectado um caso com o genótipo s1/m2.
Não se verificaram diferenças significativas na associação das variantes iceA1 e iceA2 aos diferentes tipos de CG (Quadro 3).
Quadro 3. Relação entre os vários tipos de CG e as variantes do gene iceA.
DISCUSSÃO
O primeiro objectivo deste estudo foi a aplicação de um método de detecção dos diversos genótipos de H. pylori nos carcinomas gástricos e gastrites crónicas em tecidos embebidos em parafina por PCR.
Para extracção de DNA bacteriano a partir de biopsias gástricas incluídas em parafina foram utilizados 2 métodos distintos e verificou-se que, através da técnica de extracção indicada pelo QIAamp DNA MiniKit da QIAGEN se obteve pouca quantidade de DNA, em quase todas as amostras, de um grau de pureza óptimo.
Quanto ao método de extracção de DNA de tecido sobre lâmina, obteve-se uma maior quantidade de DNA, mas não considerado de grau de pureza óptimo, devido ao facto de esta técnica não incorporar a fase de purificação do DNA. Entendeu-se então que a extracção de DNA de tecido em lâmina seria a mais indicada para este estudo, porque as biopsias gástricas incluídas em parafina são muito pequenas, dificultando a extracção e obtenção do DNA e esta técnica de extracção demonstrou que, de certo modo, se pode ultrapassar esse problema.
Também é importante referir que, se este estudo fosse realizado em estirpes de H. pylori provenientes de uma cultura in vitro, estas seriam mais favoráveis à extracção de uma maior quantidade de DNA totalmente puro, assim como promoveria a preservação da pré-selecção de estirpes e uma maior preservação dos genes bacterianos mas sem relação com o ambiente biológico de indução.
Para pesquisa dos genes cagA, vacA, iceA e suas respectivas regiões recorreu-se à nested PCR como técnica de eleição e após electroforese, observaram-se os fragmentos obtidos.
Foi procurada uma relação entre os genes estudados e a severidade das patologias em estudo. Tem sido postulado uma diversidade funcional do cagA podendo demonstrar uma relação importante com o prognóstico da doença. No nosso estudo obtivemos cagA+ em 92,3% em ambas as doenças estudadas, o que se encontra aumentado em relação a estudos anteriores, 60,4% num estudo de Figueiredo et al.27 numa população Portuguesa e 47% num estudo de González et al.28 numa população do norte de Espanha.
Uma elevada frequência de múltiplos genótipos tem sido descrito na população adulta, dependente da ecologia do H. pylori, tem sido reforçado que uma só estripe não poderá representar o pool da população bacteriana que coloniza o hospedeiro.
VacA é um importante factor de virulência na patogenese da úlcera gástrica e carcinoma gástrico. Essa toxina pode induzir várias actividades celulares, incluindo vacuolização celular, formação de canais de membrana, desregulação da função/endossomal lisossomal, apoptose e imunomodulação21. VacA tipo s1 parece ser mais virulento que o tipo s2 e tem sido associado a maior risco de gastrites atróficas e ao CG. A região vacA s2 e m2 foram os alelos mais prevalentes no CG e GC (Quadro 1), estão descritas como menos virulentos, também se verificou no estudo em infecção por H. pylori em crianças portuguesas, 67% apresentavam o genótipo de s2/m221.
Em contraste, a região vacA s1 foi mais frequente no CG do que na GC, assim como a região vacA m1, sendo considerados importantes factores de risco para o desenvolvimento do primeiro por serem considerados estripes mais virulentas.
O risco de progressão das GC para CG é elevada em doentes infectados com estripes com simultaneamente cagA+ e vacA s1/m128. No nosso estudo apenas foi encontrado em 12% (3/23) casos com este genótipo. A maioria dos GC com cagA+ estavam associadas ao vacA s2/m2 (46%,12/26). A relação das combinações s2/m2 e s1/m1 com a GC e o CG respectivamente, também foi demonstrada no estudo feito por Koehler et al., assim como, por outros estudos europeus e japoneses11,14-16.
O gene iceA tem duas variantes alélicas (iceA1 e iceA2). Segundo Yamaoka et al6 iceA1 foi o subtipo mais prevalente em uma população do leste asiático, enquanto o subtipo iceA2 foi predominante na Colômbia e nos EUA. Dados conflituantes surgem a partir de outras partes do mundo. Em uma análise de alelos iceA de H. pylori entre Finlandeses e Africanos, a presença de iceA foi significativamente menos no primeiro (35%) do que o último grupo (93%)33,34. Em outro estudo realizado na Alemanha com 141 doentes com H. pylori, iceA o gene foi detectado em 98% dos isolados de H. pylori (138 de 141)34. Resultados semelhantes foram relatados por grupos de Turquia, onde 74,7% foram positivos para iceA1 e 25,3% para iceA2, e em Xangai, onde iceA1 e iceA2 foram encontradas em 74,5% e 15,6%, respectivamente35. No nosso estudo a variante iceA1 foi a mais prevalente no GC do que em CG, com significado estatístico.
Neste estudo também foi aprofundada a presença de diferentes combinações das regiões de sinal e média do gene vacA, assim como a presença das variantes do gene iceA, consideradas factores de risco para o desenvolvimento dos vários tipos de CG. De acordo com as tabelas 2 e 3, o genótipo mais frequentemente encontrado no CG do tipo difuso foi s2/m2/iceA1, sendo considerado um importante factor de risco para o seu desenvolvimento. No CG do tipo intestinal o genótipo mais frequentemente encontrado foi s2/m2/iceA1, considerado importante factore de risco para o seu desenvolvimento. De acordo com o que já foi referido, a principal diferença entre as várias estirpes de H. pylori associadas ao CG e à GC foi a ocorrência das regiões de sinal e média do gene vacA. A combinação mais prevalente foi então s2/m2, frequentemente encontrada na GC. Este genótipo está normalmente associado a estirpes pouco virulentas, ou seja, que normalmente apresentam pouca toxicidade e que na sua maioria estão presentes nesta patologia. Logo, como se explica o facto de esta combinação ter também contribuído para o desenvolvimento da maioria dos casos de doença gástrica severa? Nas situações em que s2/m2 se encontrava associada à ao gene cagA+ e à região iceA1 pode concluir-se que provavelmente o elevado grau de patogenicidade destas estirpes se deveu à presença simultânea destes 2 factores de risco. Apenas se sabe que o aparecimento de patologias gástricas mais severas pode ser devido não só ao elevado grau de patogenicidade das estirpes do Hp, como também devido a diferenças genéticas de hospedeiro para hospedeiro e a factores ambientais32. Assim sendo, nos casos de CG em que não foi possível relacionar o genótipo com a severidade da doença, é bem possível que o seu aparecimento possa estar também ligado aos factores anteriormente mencionados.
Em geral foi possível genotipar cada estirpe bacteriana presente nas biopsias gástricas incluídas em parafina e criar uma relação de patogenicidade entre as combinações encontradas e a patologia em estudo. Apesar dos progressos que têm sido feitos, os mecanismos através dos quais infecção pelo H. pylori com estripes possuindo CagA+ ou formas activas de VacA leva ao desenvolvimento de carcinoma ainda são pouco compreendidos32.
A nested PCR foi uma óptima técnica para identificar os genes cagA, vacA e iceA em biopsias incluídas em parafina.
Estabeleceram-se parâmetros para futuras pesquisas em que seja necessário avaliar o grau da severidade da infecção gástrica provocada por diferentes estirpes o Hp. É recomendável que o estudo tenha como ponto de partida o conhecimento das várias combinações genómicas formadas pelas associações dos genes cagA, vacA, iceA e suas respectivas regiões.
Do estudo de associações entre os genótipos das estripes de H. pylori em CG e GC encontrou-se diferenças estatisticamente significativas para o alelo ice A1 (p < 0,05; OR = 4,25).
Apesar das limitações do presente estudo, a análise do vacA, cagA e iceA genes associados à virulência directamente em biopsias gástricas possibilitaram a discriminação clinicamente relevantes entre estripes de H. pylori.
REFERÊNCIAS
1. Roque AC, Pereira APG, Viegas MAP, et al. Helicobacter pylori: Aspectos Actuais da Infecção SPBS 2004;0:31-32. [ Links ]
2. Brea ML, Sanz JC, Usera MA, et al. Gastroenterítis Bacterianas, Víricas, Parasitárias Y Toxyinfecciones Alimentarias. Procedimentos em Microbiologia Clínica 1994;4:8-9. [ Links ]
3. Hofman P, Waidner B, Hofman V, et al. Pathogenesis of Helicobacter pylori infection. Gastroenterology 2004;9:1-11. [ Links ]
4. Vaira D, Holton J, Ricci C, et al. H. pylori infection from pathogenesis to treatment – a critical reappraisal. Aliment Pharmacol Ther 2002;16:105-113. [ Links ]
5. Smith MG, Hold GL, Tahara E, et al. Cellular and molecular aspects of gastric cancer. World J Gastroenterol 2006;12:2979-2990. [ Links ]
6.Qasim A, OMorain CA. Treatment of H. pylori infection and factors influencing eradication. Aliment Pharmacol Ther 2002;16:24-30. [ Links ]
7.Nogueira C, Figueiredo C, Carneiro F, et al. Helicobacter pylori Genotypes May Determine Gastric Histopathology. Am J Pathol 2001;158:647-654. [ Links ]
8. Lai CH, Kuo CH, Chen YC, et al. High prevalence of cagA and babA2-Positive H. pylori clinical isolates in Taiwan. J Clin Pathol 2002,40:3860-3862. [ Links ]
9. Höcker M, Hohenberger P. Helicobacter pylori virulence factors – one part of a big picture. Lancet 2003;362:1231-1233. [ Links ]
10. Vos M, Warren RM, Rensburg CJ, et al. Rapid genomic diversification of H. pylori. Electrofhoresis 1999;20:1476-1485. [ Links ]
11. Logan RPH, Walker MM. Epidemiology and diagnosis of H. pylori infection. BMJ 2001;323:920-922. [ Links ]
12. Go MF. Natural history and epidemiology of H. pylori infection. Aliment Pharmacol Ther 2002;16:3-15. [ Links ]
13. Oliveira AG, Santos A, Guerra JB, et al. babA2 and cagA positive H. pylori strains are associated with duodenal ulcer and gastric carcinoma in Brazil. JCM 2003,41:3964-3966. [ Links ]
14. Yoshio Y, Kodama T, Gutierrez O, et al. Relationship between Helicobacter pylori iceA, cagA and vacA status and clinical outcome: studies in four different countries JCM 1999,37:2274-2279. [ Links ]
15. Yu J, Leung WK, Go MYY, et al. Relationship between H. pylori babA2 status with gastric epithelial cell turnover and premalignant gastric lesions. Gut 2002;51:480-484. [ Links ]
16. Ikenoue T, Maeda S, Ogura K, et al. Determination of H. pylori virulence by simple gene analysis of the cag pathogenesis island. Clin Diagnostic Lab Immuno 2001;8:181-186. [ Links ]
17. Domingo D, Alarcón T, Prieto N, et al. cagA and vacA status of Spanish H. pylori clinical isolates JCM 1999;37:2113-2114. [ Links ]
18. Doorn LJ, Figueiredo C, Rossau R, et al. Typing of H. pylori vacA gene and detection of cagA gene by PCR and reverse hybridization JCM 1998;36:1271-1276. [ Links ]
19. Faundez G, Troncoso M e Figueroa G. cagA and vacA in strains of Helicobacter pylori from ulcer and non-ulcerative dyspepsia patients. BMC Gastroenterology 2002;2:20-26. [ Links ]
20. Basso D, Navaglia F, Brigato L,. et al. Analysis of H. pylori vacA and cagA genotypes and serum antibody profile in benign and malignant gastroduodenal diseases. Gut 1998;43:182-186. [ Links ]
21. Lopes AI, Palha A, Monteiro L, et al. Helicobacter pylori genotypes in children from a population at high gastric cancer risk: no association with gastroduodenal histopathology. Am J Gastroenterol 2006;101:2113-22. [ Links ]
22. Ben Mansour K, Fendri C, Zribi M, et al. Prevalence of Helicobacter pylori vacA, cagA, iceA and oipA genotypes in Tunisian patients.Ann Clin Microbiol Antimicrob 2010;19:9:10. [ Links ]
23. Ashour AAR, Collares GB, Mendes EN, et al. Magalhães PP, et al. iceA genotypes of H. pylori strains isolated from Brazilian children and adults. JCM 2001;39:1746-1750. [ Links ]
24. Xu Q, Blaser MJ. Promoters of the CATG-Specific methyl transferase gene hpyIM differ between iceA1 and iceA2 H. pylori strains. J Bacteriol 2001;183:3875-3884. [ Links ]
25. Peek RD, Doorn LJ, Donahue JP, et al. Quantitative detection of H. pylori gene expression in vivo and relationship to gastric pathology. Infection and Immun. 2000;68:5488-5495. [ Links ]
26. Gusmão VR, Mendes EN, Queiroz DMM, et al. vacA genotypes in H. pylori strains isolated from children with and without duodenal ulcer in Brazil. JCM 2000;38:2853-2857. [ Links ]
27. Figueiredo C, Van Doorn LJ, Nogueira C, et al. F.Helicobacter pylori genotypes are associated with clinical outcome in Portuguese patients and show a high prevalence of infections with multiple strains. Scand J Gastroenterol. 2001;36:128-35. [ Links ]
28. González CA, Figueiredo C, Lic CB, et al. Helicobacter pylori cagA and vacA Genotypes as Predictors of Progression of Gastric Preneoplastic Lesions: A Long-Term Follow-Up in a High-Risk Area in Spain. Am J Gastroenterol. 2011 Feb 1[Epub ahead of print]
29. Peek RD, Doorn LJ, Donahue JP, et al. Quantitative detection of H. pylori gene expression in vivo and relationship to gastric pathology. Infection and Immun 2000;68:5488-5495. [ Links ]
30. Gusmão VR, Mendes EN, Queiroz DMM, et al. vacA genotypes in H. pylori strains isolated from children with and without duodenal ulcer in Brazil JCM 2000;38:2853-2857. [ Links ]
31. Costa, A. C., Figueiredo, C. and Touati, E. Pathogenesis of Helicobacter pylori Infection. Helicobacter 2009;14:15-20. [ Links ]
32. Blaser JM, Atherton JC. Coadaptation of Helicobacter pylori and humans: ancient history, modern implications .J Clin Invest. 2009 September 1;119:2475–2487. [ Links ]
33. Karhukorpi J, Yan Y, Kolho KL, et al. cagA, vacA and iceA virulence genes of Helicobacter pylori isolates of children in Finland. Eur J Clin Microbiol Infect Dis 2000;19:790-793. [ Links ]
34. Miehlke S, Yu J, Schuppler M, et al. Helicobacter pylori vacA, iceA, and cagA status and pattern of gastritis in patients with malignant and benign gastroduodenal disease. Am J Gastroenterol 2001;96:1008-1013. [ Links ]
35. Erzin Y, Koksal V, Altun S, et al. Prevalence of Helicobacter pylori vacA, cagA, cagE, iceA, babA2 genotypes and correlation with clinical outcome in Turkish patients with dyspepsia. Helicobacter 2006;11:574-580. [ Links ]
Correspondência: Lina Carvalho; Instituto de Anatomia Patológica, Faculdade de Medicina 3000 Coimbra Email: lcarvalho@huc.min-saude.pt;
Recebido para publicação: 19/12/2008 e Aceite para publicação: 31/03/2011